近日,山东理工大学生命与医药学院姜立波教授团队与北京林业大学和瑞典于默奥大学毛建丰教授等合作团队在植物科学领域权威期刊《New Phytologist》(生物学一区TOP,IF=8.391)上发表了题为“PlantLncBoost: key features for plant lncRNA identification and significant improvement in accuracy and generalization”的文章,论文通讯作者为毛建丰教授和姜立波教授。
长链非编码RNA(lncRNA)是植物中的关键调控分子,参与基因表达调控、植物正常生长发育和环境胁迫适应等多种生物学过程。但由于lncRNA在不同物种间序列保守性较差,现有计算方法在植物lncRNA识别方面面临着泛化能力弱的问题。为解决这一难题,研究团队开发了PlantLncBoost这一新型lncRNA预测工具(图1),通过整合先进的梯度提升算法和多维度特征选择策略,显著改善了植物lncRNA识别的泛化能力。该工具通过从1662个特征中系统筛选出三个关键特征ORF覆盖度(ORF coverage)、复数傅里叶平均值(complex Fourier average)和原子傅里叶幅度(atomic Fourier amplitude),有效区分了lncRNA和mRNA。其中,ORF覆盖度是传统的序列特征,而复数傅里叶平均值和原子傅里叶幅度则是基于傅里叶变换的新型数学特征,能够捕获RNA序列的周期性和频域信息,为lncRNA识别提供了新的特征视角;通过比较CatBoost、XGBoost和LightGBM三种梯度提升算法,确定CatBoost在植物lncRNA分类任务中表现最优,这种特征与算法的结合有效改善了传统方法在跨物种应用中的局限性。
研究团队使用20个不同植物物种作为测试集,对模型进行了全面评估。测试集涵盖范围广泛,包括被子植物、苔藓植物和藻类植物等不同谱系,结果表明PlantLncBoost在所有测试物种中都保持了优异且稳定的性能(图2),证明其具有优异的跨物种泛化能力。
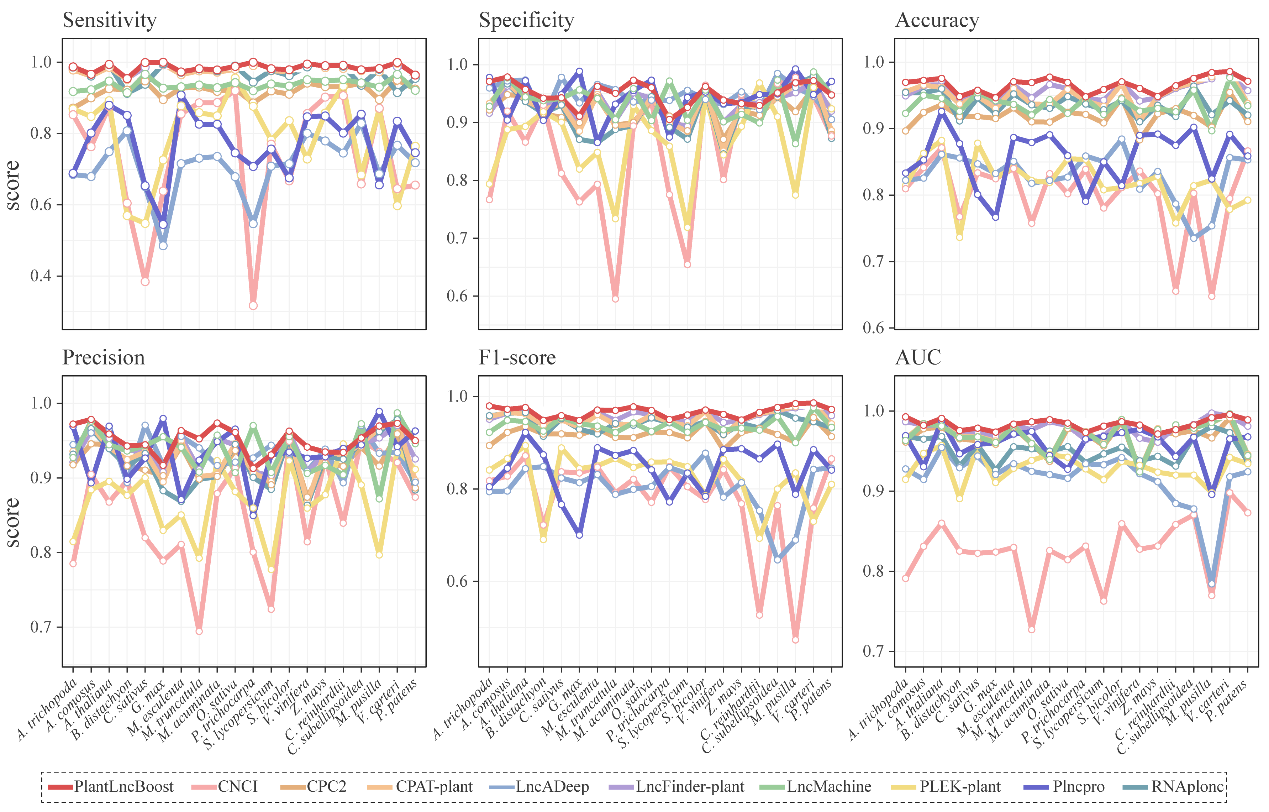
图2:10种lncRNA鉴定工具在20个植物数据集上的性能评估。
PlantLncBoost的高准确性和泛化能力将大大促进植物lncRNA的相关研究,特别是在研究较少的植物物种中发现新的lncRNA。团队已将PlantLncBoost工具开源发布在GitHub平台,地址为:https://github.com/xuechantian/PlantLncBoost。并将工具集成到综合分析流程Plant-LncRNA-pipeline v.2中(https://github.com/xuechantian/Plant-LncRNA-pipeline-v2),该流程整合了PlantLncBoost、CPAT-plant和LncFinder-plant三种lncRNA预测工具,提供了从原始数据过滤、转录组比对组装、lncRNA预测、分类到起源分析的完整工作流程,为研究者提供了一站式lncRNA分析方案,提高了植物lncRNA的研究效率。
本研究得到国家重点研发计划(2022YFD2200103)、国家自然科学基金(32171816)和山东省自然科学基金优秀青年基金(ZR2022YQ23)的支持。
论文链接:http://doi.org/10.1111/nph.70211
